Por favor, avalie esta postagem
O trabalho realizado por Peng Yin, membro do corpo docente do Wyss Core, em colaboração com Collins e outros, demonstrou que diferentes interruptores de apoio podem ser combinados para calcular a presença de múltiplos “gatilhos”, semelhante à placa lógica de um computador. Crédito: Wyss Institute da Universidade de Harvard
ADN e ARN foram comparados a “manuais de instruções” contendo as informações necessárias para o funcionamento de “máquinas” vivas. Mas enquanto as máquinas electrónicas, como os computadores e os robôs, são concebidas desde o início para servir um propósito específico, os organismos biológicos são governados por um conjunto de funções muito mais confuso e complexo, que carece da previsibilidade do código binário. Inventar novas soluções para problemas biológicos exige separar variáveis aparentemente intratáveis – uma tarefa que é assustadora até mesmo para os cérebros humanos mais intrépidos.
Duas equipes de cientistas do Instituto Wyss da Universidade de Harvard e do Instituto de Tecnologia de Massachusetts criaram caminhos para contornar esse obstáculo, indo além dos cérebros humanos; eles desenvolveram um conjunto de aprendizado de máquina algoritmos que podem analisar resmas de sequências “toehold” baseadas em RNA e prever quais serão mais eficazes na detecção e resposta a uma sequência alvo desejada. Conforme relatado em dois artigos publicados simultaneamente hoje (7 de outubro de 2020) em Comunicações da Naturezaos algoritmos também poderiam ser generalizáveis para outros problemas da biologia sintética e poderiam acelerar o desenvolvimento de ferramentas biotecnológicas para melhorar a ciência e a medicina e ajudar a salvar vidas.
“Essas conquistas são emocionantes porque marcam o ponto de partida da nossa capacidade de fazer melhores perguntas sobre os princípios fundamentais do dobramento do RNA, que precisamos conhecer para alcançar descobertas significativas e construir tecnologias biológicas úteis”, disse Luis Soenksen, Ph. D., pós-doutorado no Wyss Institute e Venture Builder na MITda Clínica Jameel, co-autor do primeiro dos dois artigos.
Nesta animação, Alex Green, Ph.D., bolsista de pós-doutorado do Wyss Institute, autor principal de “Toehold Switches: De–Novo–Designed Regulators of Gene Expression”, narra um guia passo a passo para o mecanismo do dedo do pé sintético mudar o regulador genético. Crédito: Wyss Institute da Universidade de Harvard
Obtendo interruptores de apoio
A colaboração entre cientistas de dados da Predictive BioAnalytics Initiative do Wyss Institute e biólogos sintéticos do laboratório Jim Collins, membro do corpo docente do Wyss Core, no MIT, foi criada para aplicar o poder computacional do aprendizado de máquina, redes neurais e outras arquiteturas algorítmicas a problemas complexos em biologia que até agora desafiaram a resolução. Como campo de provas para sua abordagem, as duas equipes se concentraram em uma classe específica de moléculas de RNA projetadas: interruptores de apoio, que são dobrados em forma de grampo quando estão “desligados”. Quando uma fita de RNA complementar se liga a uma sequência “gatilho” que sai de uma extremidade do grampo, a chave de apoio se desdobra em seu estado “ligado” e expõe sequências que estavam anteriormente escondidas dentro do grampo, permitindo que os ribossomos se liguem e traduzam um fluxo a jusante. gene em moléculas de proteína. Esse controle preciso sobre a expressão de genes em resposta à presença de uma determinada molécula faz com que os interruptores de posição sejam componentes muito poderosos para detectar substâncias no ambiente, detectar doenças e outros fins.
No entanto, muitos interruptores de apoio não funcionam muito bem quando testados experimentalmente, embora tenham sido projetados para produzir uma saída desejada em resposta a uma determinada entrada com base em regras conhecidas de dobramento de RNA. Reconhecendo esse problema, as equipes decidiram usar o aprendizado de máquina para analisar um grande volume de sequências de troca de apoios e usar os insights dessa análise para prever com mais precisão quais apoios executam de maneira confiável as tarefas pretendidas, o que permitiria aos pesquisadores identificar rapidamente apoios de alta qualidade para vários experimentos.
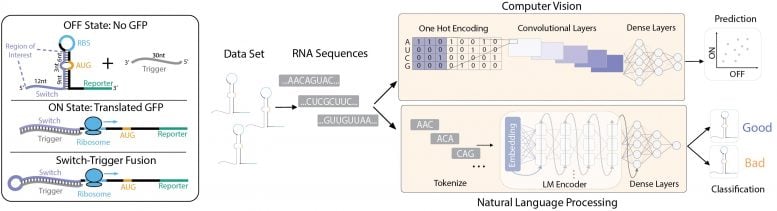
Depois de gerar um conjunto de dados de milhares de chaves de apoio, uma equipe usou um algoritmo baseado em visão computacional para analisar as sequências de apoio como imagens bidimensionais, enquanto a outra equipe usou processamento de linguagem natural para interpretar as sequências como “palavras” escritas no “linguagem” do RNA. Crédito: Wyss Institute da Universidade de Harvard
O primeiro obstáculo que enfrentaram foi que não havia um conjunto de dados de sequências de chaveamento grande o suficiente para que as técnicas de aprendizado profundo fossem analisadas de maneira eficaz. Os autores se encarregaram de gerar um conjunto de dados que seria útil para treinar tais modelos. “Projetamos e sintetizamos uma enorme biblioteca de interruptores de apoio, quase 100.000 no total, amostrando sistematicamente regiões de gatilho curtas ao longo de todo o genoma de 23 vírus e 906 fatores de transcrição humanos”, disse Alex Garruss, estudante de graduação de Harvard que trabalha no Wyss Institute. que é co-autor do primeiro artigo. “A escala sem precedentes deste conjunto de dados permite o uso de técnicas avançadas de aprendizado de máquina para identificar e compreender switches úteis para aplicações downstream imediatas e projetos futuros.”
Munidas de dados suficientes, as equipes primeiro empregaram ferramentas tradicionalmente usadas para analisar moléculas sintéticas de RNA para ver se conseguiam prever com precisão o comportamento dos interruptores de apoio, agora que havia muitos mais exemplos disponíveis. No entanto, nenhum dos métodos que tentaram — incluindo a modelação mecanicista baseada na termodinâmica e nas características físicas — foi capaz de prever com suficiente precisão. precisão quais apoios para os pés funcionaram melhor.
Uma imagem vale mil pares de bases
Os pesquisadores então exploraram várias técnicas de aprendizado de máquina para ver se poderiam criar modelos com melhores habilidades preditivas. Os autores do primeiro artigo decidiram analisar os toehold switches não como sequências de bases, mas sim como “imagens” bidimensionais de possibilidades de pares de bases. “Conhecemos as regras básicas de como os pares de bases de uma molécula de RNA se ligam uns aos outros, mas as moléculas são onduladas – elas nunca têm uma única forma perfeita, mas sim uma probabilidade de diferentes formas em que poderiam ter”, disse Nicolaas Angenent-Mari, um estudante de pós-graduação do MIT que trabalha no Wyss Institute e co-autor do primeiro artigo. “Algoritmos de visão computacional tornaram-se muito bons na análise de imagens, então criamos uma representação semelhante a uma imagem de todos os possíveis estados de dobramento de cada chave de apoio e treinamos um algoritmo de aprendizado de máquina nessas imagens para que pudesse reconhecer os padrões sutis indicando se um determinada imagem seria um apoio bom ou ruim.”
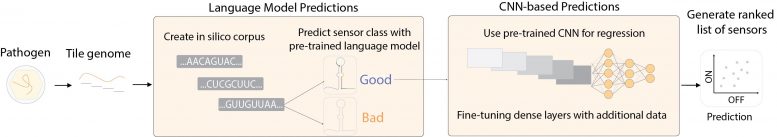
Ao usar ambos os modelos sequencialmente, os pesquisadores foram capazes de prever quais sequências de apoio produziriam sensores de alta qualidade. Crédito: Wyss Institute da Universidade de Harvard
Outro benefício de sua abordagem baseada no visual é que a equipe foi capaz de “ver” quais partes de uma sequência de chave de apoio o algoritmo “prestou mais atenção” ao determinar se uma determinada sequência era “boa” ou “ruim”. Eles chamaram essa abordagem de interpretação de Visualização de mapas de saliência de estrutura secundária, ou VIS4Map, e a aplicaram a todo o conjunto de dados do switch de apoio. O VIS4Map identificou com sucesso os elementos físicos dos interruptores de apoio que influenciaram seu desempenho e permitiu aos pesquisadores concluir que os apoios com estruturas internas potencialmente mais concorrentes eram “mais vazados” e, portanto, de qualidade inferior do que aqueles com menos estruturas desse tipo, fornecendo informações sobre mecanismos de dobramento de RNA que não foram descobertos usando técnicas de análise tradicionais.
“Ser capaz de compreender e explicar por que certas ferramentas funcionam ou não tem sido um objetivo secundário dentro da comunidade de inteligência artificial há algum tempo, mas a interpretabilidade precisa estar na vanguarda das nossas preocupações quando estudamos biologia porque as razões subjacentes para essas o comportamento dos sistemas muitas vezes não pode ser intuído”, disse Jim Collins, Ph.D., autor sênior do primeiro artigo. “Descobertas e perturbações significativas são o resultado de uma compreensão profunda de como a natureza funciona, e este projeto demonstra que a aprendizagem automática, quando devidamente concebida e aplicada, pode melhorar significativamente a nossa capacidade de obter conhecimentos importantes sobre sistemas biológicos.” Collins também é Professor Termeer de Engenharia Médica e Ciência no MIT.
Agora você está falando minha língua
Enquanto a primeira equipe analisou sequências de chaveamento como imagens 2D para prever sua qualidade, a segunda equipe criou duas arquiteturas diferentes de aprendizagem profunda que abordaram o desafio usando técnicas ortogonais. Eles então foram além da previsão da qualidade do apoio e usaram seus modelos para otimizar e redesenhar interruptores de apoio de baixo desempenho para diferentes finalidades, o que eles relatam no segundo artigo.
O primeiro modelo, baseado em uma rede neural convolucional (CNN) e perceptron multicamadas (MLP), trata sequências de toehold como imagens 1D, ou linhas de bases de nucleotídeos, e identifica padrões de bases e interações potenciais entre essas bases para prever boas e pés ruins. A equipe usou esse modelo para criar um método de otimização chamado STORM (Sequence-based Toehold Optimization and Redesign Model), que permite o redesenho completo de uma sequência de apoio desde o início. Esta ferramenta de “folha em branco” é ideal para gerar novos interruptores de apoio para desempenhar uma função específica como parte de um circuito genético sintético, permitindo a criação de ferramentas biológicas complexas.
“A parte realmente legal do STORM e do modelo subjacente é que, depois de semeá-lo com os dados de entrada do primeiro artigo, conseguimos ajustar o modelo com apenas 168 amostras e usar o modelo aprimorado para otimizar os interruptores de apoio. Isso questiona a suposição predominante de que é necessário gerar conjuntos de dados massivos toda vez que quiser aplicar um algoritmo de aprendizado de máquina a um novo problema e sugere que o aprendizado profundo é potencialmente mais aplicável para biólogos sintéticos do que pensávamos”, disse o co-primeiro autora Jackie Valeri, estudante de pós-graduação do MIT e do Wyss Institute.
O segundo modelo é baseado no processamento de linguagem natural (PNL) e trata cada sequência de apoio como uma “frase” que consiste em padrões de “palavras”, aprendendo eventualmente como certas palavras são reunidas para formar uma frase coerente. “Gosto de pensar em cada mudança de posição como um poema haicai: como um haicai, é um arranjo muito específico de frases em sua língua mãe – neste caso, RNA. Estamos essencialmente treinando esse modelo para aprender como escrever um bom haicai, alimentando-o com muitos e muitos exemplos”, disse o coautor Pradeep Ramesh, Ph.D., pesquisador visitante de pós-doutorado no Wyss Institute e cientista de aprendizado de máquina no Sherlock Biociências.
Ramesh e seus coautores integraram este modelo baseado em PNL com o modelo baseado em CNN para criar NuSpeak (Nucleic Acid Speech), uma abordagem de otimização que lhes permitiu redesenhar os últimos 9 nucleotídeos de um determinado toehold switch, mantendo os 21 nucleotídeos restantes. intacto. Esta técnica permite a criação de bases projetadas para detectar a presença de sequências específicas de RNA patogênico e pode ser usada para desenvolver novos testes de diagnóstico.
A equipe validou experimentalmente ambas as plataformas otimizando interruptores de apoio projetados para detectar fragmentos do SARS-CoV-2 genoma viral. O NuSpeak melhorou o desempenho dos sensores em uma média de 160%, enquanto o STORM criou versões melhores de quatro sensores de RNA viral SARS-CoV-2 “ruins”, cujo desempenho melhorou em até 28 vezes.
“Um benefício real das plataformas STORM e NuSpeak é que elas permitem projetar e otimizar rapidamente componentes de biologia sintética, como mostramos com o desenvolvimento de sensores de apoio para dedos para um COVID 19 diagnóstico”, disse a co-primeira autora Katie Collins, uma estudante de graduação do MIT no Wyss Institute que trabalhou com o professor associado do MIT Timothy Lu, MD, Ph.D., autor correspondente do segundo artigo.
“As abordagens baseadas em dados possibilitadas pelo aprendizado de máquina abrem a porta para sinergias realmente valiosas entre a ciência da computação e a biologia sintética, e estamos apenas começando a arranhar a superfície”, disse Diogo Camacho, Ph.D., autor correspondente do segundo artigo, que é Cientista Sênior de Bioinformática e co-líder da Predictive BioAnalytics Initiative no Wyss Institute. “Talvez o aspecto mais importante das ferramentas que desenvolvemos nestes artigos é que elas são generalizáveis para outros tipos de sequências baseadas em RNA, como promotores indutíveis e riboswitches de ocorrência natural e, portanto, podem ser aplicadas a uma ampla gama de problemas e oportunidades em biotecnologia e medicina.”
Autores adicionais dos artigos incluem membro do corpo docente do Wyss Core e professor de genética no HMS George Church, Ph.D.; e os alunos de pós-graduação da Wyss e do MIT Miguel Alcantar e Bianca Lepe.
“A inteligência artificial é uma onda que está apenas começando a impactar a ciência e a indústria e tem um potencial incrível para ajudar a resolver problemas intratáveis. Os avanços descritos nestes estudos demonstram o poder da fusão da computação com a biologia sintética na bancada para desenvolver novas e mais poderosas tecnologias bioinspiradas, além de levar a novos insights sobre mecanismos fundamentais de controle biológico”, disse Don Ingber, MD, Ph. D., Diretor Fundador do Wyss Institute. Ingber também é professor Judah Folkman de Biologia Vascular na Harvard Medical School e do Programa de Biologia Vascular no Boston Children's Hospital, bem como professor de Bioengenharia na Escola de Engenharia e Ciências Aplicadas John A. Paulson de Harvard.
Referências:
“Uma abordagem de aprendizagem profunda para interruptores de RNA programáveis” por Nicolaas M. Angenent-Mari, Alexander S. Garruss, Luis R. Soenksen, George Church e James J. Collins, 7 de outubro de 2020, Comunicações da Natureza.
DOI: 10.1038/s41467-020-18677-1
“Estruturas de aprendizagem profunda de sequência para função para riborreguladores projetados” por Jacqueline A. Valeri, Katherine M. Collins, Pradeep Ramesh, Miguel A. Alcantar, Bianca A. Lepe, Timothy K. Lu e Diogo M. Camacho, 7 de outubro de 2020 , Comunicações da Natureza.
DOI: 10.1038/s41467-020-18676-2
Este trabalho foi apoiado pela DARPA Programa de descoberta e design sinérgico, a Agência de Redução de Ameaças de Defesa, o Paul G. Allen Frontiers Group, o Instituto Wyss de Engenharia Biologicamente Inspirada, a Universidade de Harvard, o Instituto de Engenharia Médica e Ciência, o Instituto de Tecnologia de Massachusetts, a Fundação Nacional de Ciência, o Instituto Nacional de Pesquisa do Genoma Humano, o Departamento de Energia, o Instituto Nacional de Saúdee uma bolsa CONACyT.
