Por favor, avalie esta postagem
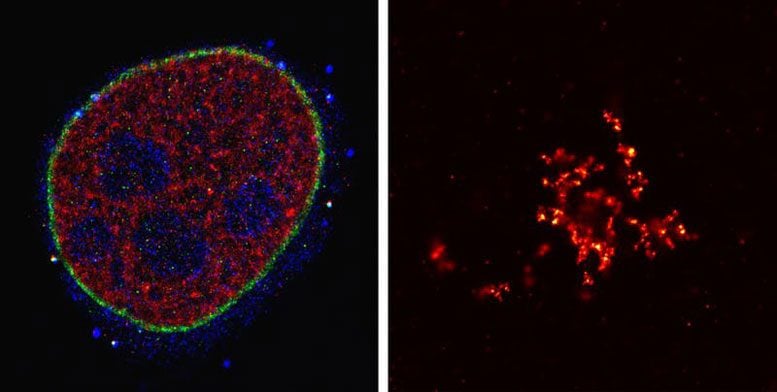
Os pesquisadores usaram o método SDC-PAINT para visualizar a rede de filamentos de microtúbulos do citoesqueleto (verde) e sua proximidade com duas proteínas adicionais chamadas TOM20 (vermelho) e HSP60 (azul). Cada imagem mostra as proteínas em um plano diferente da célula começando de cima, e as imagens ampliadas na parte inferior comparam a resolução obtida com SDC-PAINT (esquerda) com a possível com microscopia confocal convencional (direita). Crédito: Florian Schueder, MPI/LMU
Os biólogos celulares tradicionalmente usam corantes fluorescentes para rotular e visualizar células e as moléculas dentro delas sob um microscópio. Com diferentes métodos de microscopia de super-resolução, eles podem até iluminar moléculas únicas e ver suas interações complexas entre si. No entanto, o hardware de microscopia necessário para fazer isso é altamente especializado e caro, e exige que os operadores tenham habilidades únicas; portanto, tais microscópios são relativamente raros em laboratórios de todo o mundo.
Ralf Jungmann, Ph.D., ex-aluno do Wyss Institute e atualmente professor de Bioquímica na Ludwig Maximilian University (LMU) e no Max Planck Institute (MPI) na Alemanha e membro do corpo docente do Wyss Institute Peng Yin, Ph.D. . têm vindo a desenvolver ADN-PAINT, uma poderosa tecnologia de imagem molecular que envolve interações transitórias de DNA-DNA para localizar com precisão corantes fluorescentes com super-resolução. No entanto, embora os investigadores tenham demonstrado o potencial do DNA-PAINT ao visualizar biomoléculas únicas, como proteínas, em células fixas a uma distância fixa e próxima, a tecnologia ainda não conseguiu investigar moléculas nas profundezas das células.
Agora, as equipes de Jungmann e Yin relatam em conjunto uma solução para superar essa limitação. Em seu novo estudo, eles adaptaram a tecnologia DNA-PAINT para microscópios confocal, que são amplamente utilizados por pesquisadores em laboratórios de biologia celular para obter imagens de células inteiras e tecidos mais espessos em resolução mais baixa. A equipe do MPI/Wyss Institute demonstra que o método pode visualizar uma variedade de moléculas diferentes, incluindo combinações de diferentes proteínas, RNAs e DNA em toda a profundidade de células inteiras em super-resolução. Publicado em Comunicações da Naturezaa abordagem poderia abrir a porta para estudos detalhados de localização de moléculas únicas em muitas áreas da pesquisa celular.
A abordagem DNA-PAINT anexa uma “fita âncora” de DNA à molécula de interesse. Em seguida, uma “cadeia de imagem” de DNA marcada com corante com uma sequência complementar se liga transitoriamente à âncora e produz um sinal fluorescente, que ocorre como um evento intermitente definido em locais moleculares únicos. Como esta “frequência intermitente” é precisamente ajustável, moléculas que estão separadas apenas por nanômetros umas das outras podem ser distinguidas – no final de resolução mais alta da super-resolução.
“Nossa nova abordagem, SDC-PAINT, integra os versáteis recursos de super-resolução do DNA-PAINT com os recursos de corte óptico dos microscópios confocal. Assim, criamos os meios para explorar toda a profundidade de uma célula e visualizar as moléculas dentro dela em escala nanométrica”, disse Jungmann. A equipe mapeou a presença de diferentes combinações de proteínas em células inteiras e foi além disso. “Ao diversificar nossas abordagens de rotulagem, também visualizamos diferentes tipos de biomoléculas individuais no núcleo que contém o cromossomo, incluindo sequências no DNA, proteínas ligadas ao DNA ou à membrana que envolve o núcleo, bem como RNAs nucleares”, acrescenta Yin, que também é colíder da Iniciativa de Robótica Molecular do Wyss Institute e professor de Biologia de Sistemas na Harvard Medical School.
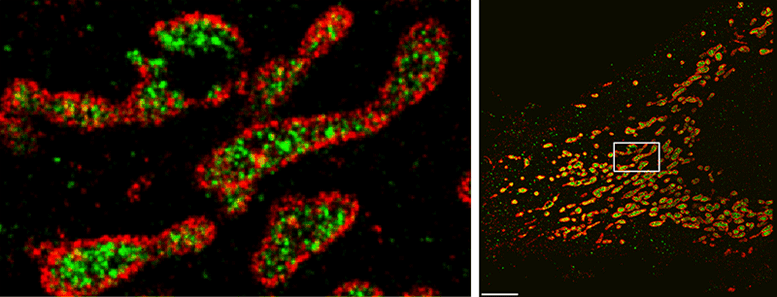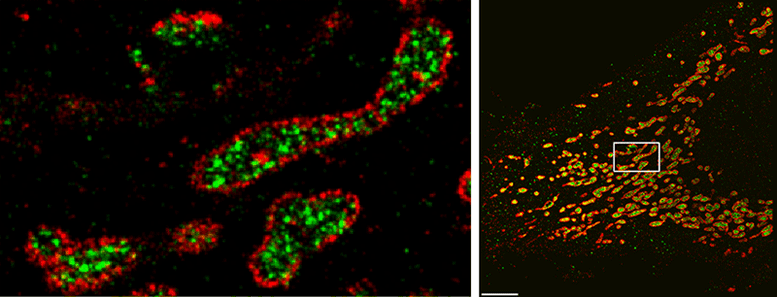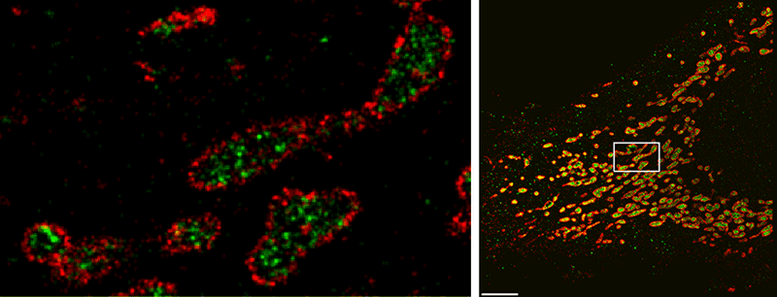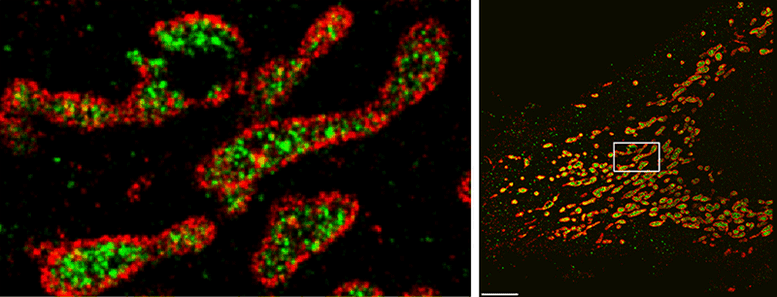
O SDC-PAINT pode visualizar com precisão a distribuição das estruturas produtoras de energia da célula, conhecidas como mitocôndrias, combinando sinais de fluorescência de alta resolução de uma proteína em suas membranas externas (em vermelho) e uma proteína em seu lúmen interno (em verde). O Gif à esquerda mostra seções consecutivas tiradas do espaço 3D das células na área delimitada pelo pequeno retângulo na imagem à direita. Crédito: Florian Schueder, MPI/LMU
Em princípio, os microscópios confocal usam os chamados furos para eliminar a fluorescência fora de foco indesejada dos planos de imagem acima e abaixo do plano focal. Ao examinar a amostra, plano após plano, os pesquisadores podem reunir os sinais de fluorescência desejados emitidos pelos corantes ligados às moléculas em toda a profundidade. Especificamente, a equipe do MPI/Wyss Institute desenvolveu a técnica para microscópios “Spinning Disk Confocal” (SDC) que detectam sinais de fluorescência de um plano inteiro de uma só vez, detectando-os através de um disco giratório com vários orifícios. Além disso, “para alcançar a super-resolução 3D, colocamos uma lente adicional no caminho de detecção, o que nos permite arquivar a resolução limitada por subdifração na terceira dimensão”, disse o primeiro autor Florian Schueder, um estudante de pós-graduação que trabalha com Jungmann que também trabalhou com a equipe do Yin’s Wyss Institute como parte de sua tese de mestrado.
“Esta adição pode ser facilmente personalizada pelos fabricantes de microscópios SDC; então basicamente implementamos microscopia de super-resolução sem alterações complexas de hardware em microscópios que geralmente estão disponíveis para biólogos celulares de todos os locais de pesquisa biomédica. A abordagem tem, portanto, o potencial de democratizar imagens de super-resolução de células e tecidos inteiros”, disse Jungmann.
A imagem da esquerda mostra como os pesquisadores usaram o SDC-PAINT para investigar o núcleo que envolve o DNA de uma célula com a membrana nuclear em verde, uma proteína associada ao cromossomo em vermelho e uma proteína que controla diferentes processos nucleares em azul. Na imagem à direita, a localização de um RNA específico chamado Xist em diferentes regiões do cromossomo X é demonstrada com a abordagem. Crédito: Brian Beliveau, Sinem Saka, Hiroshi Sasaki, Wyss Institute da Universidade de Harvard
“Com este importante avanço, a microscopia de super-resolução e o DNA-PAINT poderiam se tornar mais acessíveis aos pesquisadores biomédicos, acelerando nossos insights sobre a função de moléculas individuais e os processos que elas controlam dentro das células”, disse o diretor fundador do Wyss Institute, Donald Ingber, MD, Ph.D., que também é professor Judah Folkman de Biologia Vascular no HMS e do Programa de Biologia Vascular no Hospital Infantil de Boston, bem como professor de Bioengenharia na Escola de Engenharia e Ciências Aplicadas (SEAS) John A. Paulson de Harvard.
Outros autores do estudo são membros antigos e atuais do grupo de Yin, incluindo Juanita Lara-Gutiérrez; Brian Beliveau, Ph.D.; Sinem Saka, Ph.D.; e Hiroshi Sasaki, Ph.D.; e Johannes Woehrstein, Maximilian Strauss e Heinrich Grabmayr, Ph.D., que estão trabalhando com Jungmann. O estudo foi financiado por doações do Instituto Wyss de Engenharia Biologicamente Inspirada da Universidade de Harvard, do Programa Emmy Noether da Fundação Alemã de Pesquisa, do Conselho Europeu de Pesquisa, do Centro de Nanociência da LMU, da Sociedade Max Planck e da Fundação Max Planck, da Instituto Nacional de Saúde e o Escritório de Pesquisa Naval.
Referência: “Imagem de super-resolução 3D multiplexada de células inteiras usando microscopia confocal de disco giratório e DNA-PAINT” por Florian Schueder, Juanita Lara-Gutiérrez, Brian J. Beliveau, Sinem K. Saka, Hiroshi M. Sasaki, Johannes B. Woehrstein , Maximilian T. Strauss, Heinrich Grabmayr, Peng Yin e Ralf Jungmann, 12 de dezembro de 2017, Comunicações da Natureza.
